梭状芽孢杆菌是一个多样化的革兰氏阳性菌属,专性厌氧,在环境中普遍存在。这个属大约包含了100多个种,基因组内总体的G+C含量范围在22-55%,反映出该属内的细菌在系统进化关系上的巨大差异。
该属内最主要的食源性致病菌为肉毒梭菌和产气荚膜梭菌,它们均可通过毒素介导,通过直接产生毒素(如食源性肉毒中毒),或在肠道内生成毒素(如婴儿肉毒中毒和产气荚膜梭菌腹泻),从而引发食源性细菌性疾病。
肉毒梭菌简介
肉毒梭菌(Clostridium botulinum)属于厌氧性梭状芽孢杆菌,为革兰氏阳性的多形态大杆菌,芽孢位于菌体近端,使菌体呈匙形或网球拍状。根据抗原性的不同,肉毒梭菌可分为8个型,分别是A、B、Cα、Cβ、D、E、F和G型。
肉毒梭菌产生的外毒素称为肉毒神经毒素(BoNTs,botulinum neurotoxins),肉毒毒素在目前已知毒素中毒性最强,一个人的致死剂量大约为1 µg,比氰化钾毒力大10000倍,和炭疽一样,可作为生物武器。BoNTs可引发食物中毒,即肉毒中毒,能够引起人类中毒的主要型别为A、B和E型。肉毒中毒的临床表现不同于其他食物中毒,胃肠道症状很少见,主要为神经末梢麻痹,发病率不高,但死亡率居细菌性食物中毒之首。
肉毒梭菌是一种腐物寄生菌,在自然界分布广泛,食物中肉毒梭菌主要来源于环境,如尘埃、粪便等,尤其是带菌土壤。肉毒梭菌的抵抗力一般,在45℃以上都受到抑制,80℃经20min可被杀灭。但其芽孢的抵抗力极强,可耐煮沸长达1-6h之久,于180℃干热5-15min或于120℃高压蒸汽下10-20min才能灭活,在酒精中可存活2个月,10%的HCl需60min才能破坏芽孢。蛋白分解型的肉毒梭菌(A、B和F型)芽孢比非蛋白分解型的B、E和F型的芽孢对热具有更强的抵抗力。芽孢在厌氧环境的食品中可发芽繁殖产生肉毒毒素,人类因食入已产生毒素的食品而发生肉毒中毒。另外,因肉毒梭菌随蜂蜜等食物进入婴儿肠道内产毒引发的婴儿肉毒中毒也有存在。
肉毒梭菌的形态特征和分类学
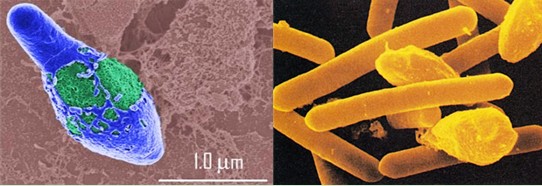
肉毒梭菌为多形态的厌氧性的杆状菌,长约4-6?m,宽约0.6-1.2 ?m,两侧平行,两端钝圆,直杆状或稍弯曲,可形成芽孢,卵圆形,位于次级端或偶有位于中央,常见很多游离芽孢,多单在,偶见成双或短链,A型和B型菌的芽孢大于菌体,位于菌体近端,使菌体呈匙形或网球拍状,其他的型芽孢一般不超过菌体宽度。有时呈现长丝状或链状,有时能见到舟形、带把柄的柠檬形、蛇样线状和染色较深的球茎状,这些属于退化型,当菌体开始形成芽孢时,常常伴随着自溶现象。肉毒梭菌具有4-8根周生性鞭毛,运动迟缓,没有荚膜。
根据《伯杰氏细菌鉴定手册》,肉毒梭菌归属为原核生物界、厚壁菌门、厚壁菌纲、芽孢杆菌科的梭菌属(Clostridium)。
肉毒梭菌这个种是基于单一的表型特征,即产生BoNTs的这个特性来分类定义的。Collins等(1998)又将其分为4个组(Group I-IV),这些群呈现出非常多样化的基因型和表型特征(见表一),菌株之间的差异程度要远远超过枯草芽孢杆菌(Bacillus subtilis)和金黄色葡萄球菌(Staphylococcus aureus)菌株之间的差异程度。
这种情况有时更为复杂,导致分类学上的困难,因为有些菌株从发育关系上来说与肉毒梭菌非常相近,但是并不产生神经毒素,因而没有归入肉毒梭菌这个种,如生孢梭菌(Clostridium sporogenes),诺维氏梭菌(Clostridium novyi),和其他一些未命名的梭菌;而有些菌株能生产神经毒素,却因系统发育较远,并未归入肉毒梭菌,如巴氏梭菌(Clostridium baratii)以及酪酸梭菌(Clostridium butyricum)。更复杂的情况是,Collins等(1998)还发现BoNTs的毒力基因能在菌株间不稳定地平行转移,从而产生不产毒的变种,或是携带部分或未知的BoNTs基因。正因为肉毒梭菌的种内多样性,导致了对于该细菌的分类学研究仍存在争议,给各种肉毒梭菌的检测、分离和鉴定方法,甚至是16S测序法鉴定都带来了一定的难度。
表一:肉毒梭菌的基本特征
特征 | 组I | 组II | 组III | 组IV |
毒素类型 | A, B, F | B, E, F | C, D | G |
人类疾病病原 | 是 | 是 | 是 | 否 |
最低生长温度(℃) | 10 | 3.3 | 15 | 不确定 |
最适生长温度(℃) | 35-40 | 18-25 | 40 | 37 |
最低生长pH值 | 4.6 | 5.0 | 5.0 | 不确定 |
最低生长水活度Aw | 0.94 | 0.97 | 不确定 | 不确定 |
氯化钠抑制 | 10% | 5% | 2% | 不确定 |
芽孢D100℃值(分钟) | 25 | < 0.1 | 0.1-0.9 | 0.8-1.12 |
芽孢D121℃值(分钟) | 0.1-0.2 | < 0.001 | 不确定 | 不确定 |
蛋白分解活性 | 是 | 否 | 否 | 微弱 |
脂肪分解活性 | 是 | 是 | 是 | 否 |
糖分解活性 | 否 | 是 | 微弱 | 否 |
相近的非产毒梭菌属菌种 | C. sporogenes C. putrificum | C. beijerinkii | C. novyi C. haemolyticum | C. subterminale C. histolyticum C. linosium |
毒力基因位置 | 染色体 | 染色体 | 噬菌体 | 质粒 |
基因组G+C含量(mol%) | 26-29 | 27-29 | 26-28 | 28-30 |
肉毒梭菌的传统鉴定和分离方法
正如前述,肉毒梭菌这个种是基于单一的表型特征,即产生BoNTs的这个特性来分类定义的,所以传统的表型和基因型鉴定方法,如生化反应、细胞脂肪酸组分分析以及16S测序等并不能给出明确的鉴定结果,见Collins(1998),Brett(1998),Ghanem(1991)。比如,16S测序方法已被证实无法区分肉毒梭菌及其两个近亲:诺维氏梭菌(Clostridium novyi)和生孢梭菌(Clostridium sporogenes)。
肉毒梭菌培养分离的第一步是增菌培养基及培养条件的确定。培养基可优先选用预还原性(Pre-reduced)厌氧无菌的熟肉基质培养基(可含或不含葡萄糖成分)(cooked meat medium),碎肉葡萄糖淀粉培养基(chopped-meat glucose-starch medium),或胰蛋白胨葡萄糖酵母提取肉汤(tryptone-peptone glucose yeast extract broth)。胰蛋白酶可被添加到配方中以灭活细菌素和激发神经毒素。溶菌酶的加入有助于复活热损伤的孢子,尤其适合于非蛋白分解型肉毒梭菌的菌株。
由于不同组别的肉毒梭菌的最适生长条件并不统一(见表一),所以最优的培养温度的选择仍然是个问题。Solomon(2001)等推荐采用28℃用于II组肉毒梭菌的培养,而用35℃用于其他组别的培养,不过目前已经有质疑认为其不适用鱼类和贝壳类食品中肉毒梭菌的检测。Anon(1998)等推荐采用30℃用于所有组别的菌株的培养,但在这个条件下I组的菌株得不到最优的生长。
培养5天后,接种的肉汤需做小鼠生物实验(Mouse Bioassay)以检测BoNTs的产生情况。另需培养10天以检测受损芽孢或延迟产芽菌株的生长情况仍然是十分必要的。所有的培养物还需最终通过镜检检测,观察菌株的形态特征,以获得鉴定结论。
疑似阳性的肉汤样本,连同疑似原因食品和临床样本(尤其是粪便)还需选用上述培养基进行严格厌氧环境下的二次培养以进行确认。培养基需包含以下成分:
卵黄:以检测酯酶阳性的肉毒梭菌菌株,见Silas(1985)
选择性抗菌剂:环丝氨酸,磺胺甲基异恶唑,甲氧苄啶等,见Silas(1985),Dezfulian(1981),Mills(1985)
由于肉毒梭菌不同组别菌株存在较大差异(尤其是I组和II组),专家们一致建议采用含抑菌剂和不含有抑菌剂的选择性平板同时进行对照实验,以便分析和获得更为准确的结果。
典型的肉毒梭菌的菌落形态见图一:
Typical colonial growth of C. botulinum, together with an atypical lipase negative culture: Classically, C. botulinum colonies are usually gray-white on blood containing agars with circular irregular edges, and a clear zone of β-hemolysis.

Clostridium botulinum growing on blood agar (Below Left)
Clostridium botulinum growing on egg yolk agar showing the lipase reaction after 72 hours of incubation (Below Right)

主要的肉毒梭菌的生化特征见表二和表三:
表二:各型肉毒梭菌的生化特性
型别 | A | B | C | D | E | F |
葡萄糖发酵 | + | + | + | + | + | + |
麦芽糖发酵 | + | (±) | (±) | (±) | (±) | (±) |
乳糖发酵 | - | - | (-) | (-) | - | - |
蔗糖发酵 | (-) | (±) | - | - | - | (+) |
靛基质产生 | (±) | - | (-) | (-) | - | - |
明胶液化 | - | (+) | (±) | (±) | (±) | (+) |
牛奶消化 | (+) | (±) | - | - | - | (+) |
注:肉毒梭菌的生化形状并不规律,即使同型也常见到株间差异;+表示阳性,-表示阴性,(±)表示视菌株而定,(+)表示多为阳性反应,(-)表示多为阴性反应。
表三:各组肉毒梭菌的生化特性
组别 | I组 | II组 | III组 | IV组 | |||||||
芽孢位置 | S | T | S | T | S | T | S | T | |||
动力 | + | - | + | + | - | + | |||||
有氧环境 | - | - | - | - | |||||||
葡萄糖 | + | + | + | - | |||||||
乳糖 | - | - | - | - | |||||||
麦芽糖 | - | + | d | - | |||||||
甘露醇 | - | - | - | - | |||||||
卵磷脂酶 | - | - | - | + | - | ||||||
酯酶 | + | + | + | - | |||||||
七叶苷水解 | + | - | - | - | |||||||
肉渣消化 | + | - | + | - | + | - | |||||
明胶液化 | + | + | + | + | |||||||
吲哚产生 | - | - | - | + | - | ||||||
牛乳反应 | d | c | - | d | d | ||||||
硝酸盐还原 | - | - | - | - | |||||||
脲酶 | - | - | - | - | |||||||
肉毒梭菌的Ribotyping鉴定及分子分型
正因为肉毒梭菌的种内菌株的多样性及其16S序列的高度保守性使得生化鉴定和16S测序鉴定等传统技术对肉毒梭菌的鉴定存在诸多难点,所以研究者们不遗余力应用更多分子生物学技术以缩短肉毒梭菌的鉴定时间,简化鉴定流程,增加鉴定结果的准确性和重现性。
Ribotyping(核糖体分型)是首个用于细菌鉴定和分型的分子指纹技术,无需依赖PCR技术,因此可避免非特异扩增问题,具有较高的保真性。其是在RFLP(限制性片段长度多态性)和Southern Blot印迹基础上发展起来的一种鉴定和分型方法,它将细菌的全基因组DNA用限制性内切酶消化,经电泳分离和Southern Blot转印后,用放射性标记的rRNA操纵子探针杂交,根据带型和带数的多态性对细菌进行分子分型,同时因图谱本身具备系统分类学的意义,因此可将图谱与标准菌株数据库的指纹模式(Ribo Pattern)进行比对,实现鉴定和分子分型的同步完成。
该方法的优点之一是广谱性、通用性和特异性兼顾。由于核糖体编码基因有较高的保守性,各种限制性酶切位点在细菌基因组内也广泛分布,因此可用一种通用的探针和酶切方案对多种不同的微生物进行检测,实现广谱分析,特别适合未知病原菌的筛查和溯源,也满足大规模检测及食物中毒应急检测的需求。用户也可为某一特定种属的细菌进行酶切方案和探针的探索及优化,选用单一酶切或混和酶切的条件,及更为特异的探针,满足更高特异性和分型力的需求,建立个性化的菌株指纹图谱数据库,见Grimont(1986)。
该方法的另一大优点是分型力较好。由于核糖体是细菌体内重要的蛋白质合成“工厂”,细菌通常具有多个核糖体编码基因以满足新陈代谢需要。通过Ribotyping,可获得足够数量不同分子大小的指纹条带(一般为5~15条),具备较高的分型率和良好的重复性,分型图谱稳定性高、条带多,见Anne(1991)。
Schalch等(1997)曾采用Ribotyping技术对10个食物中毒暴发事件中收集而来的34株产气荚膜梭菌(Clostridium perfringens)进行了分型,得到了12种指纹模式(Ribotype),且从原因食品中和病人粪便样本中分离得到的菌株的指纹模式高度吻合。
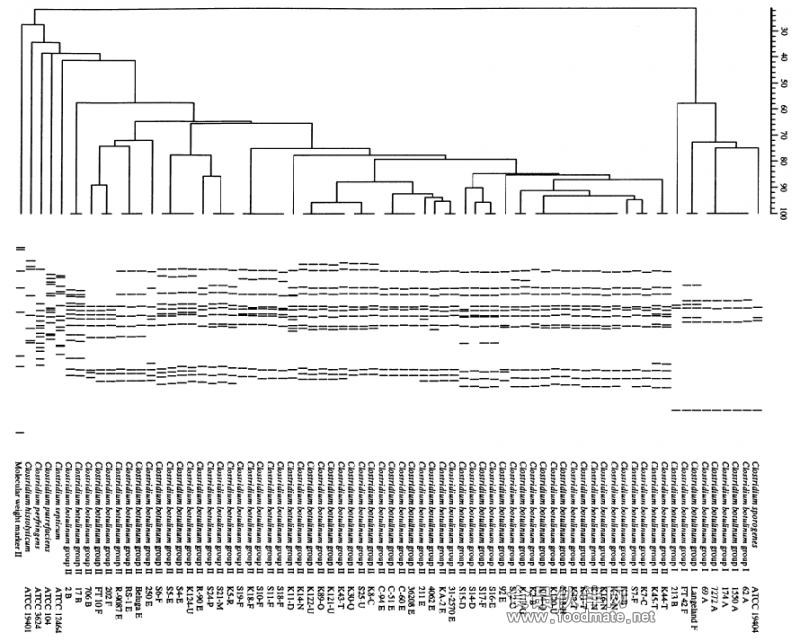
Hielm等(1999)首次采用Ribotyping技术对68株肉毒梭菌和5种相关的梭菌属菌种进行了鉴定和分型,以考察该技术对引发中毒的肉毒梭菌的鉴定和分型能力。他们先后试验了13种限制性内切酶,最终选择以EcoR I和Hind III的酶切方案进行鉴定和分型。这两种酶切方案及Ribotyping技术均显示出了良好的鉴定和分型效果,可以显著的区分蛋白分解型和非蛋白分解型的肉毒梭菌菌株,并被推荐为肉毒梭菌种属鉴定的优选方法。
他们随后用GelCompar软件对两种酶切方案得到的指纹图谱进行了分型力分析以及UPGMA分析。结果显示(见表四、图二和图三),EcoR I酶切获得的Ribotyping指纹图谱分型效果最好,分型指数(Discriminatory Index)高达0.982;Hind III酶切的分型能力也相当,分型指数为0.954。聚类分析的结果显示,各菌株间的Dice相似系数为35±13%(中值±标准差)。这些结果都表明Ribotyping技术非常适合用于肉毒梭菌的鉴定和分子分型。
Hielm等还认为,对于肉毒梭菌,Ribotyping的分型能力与PFGE(脉冲场凝胶电泳)相当,且其图谱具备PFGE所缺乏的细菌分类学意义,因此可用于肉毒梭菌的鉴定,而PFGE图谱则因其随机酶切的性质,无法用于鉴定。另一方面,随着自动化的Ribotyping技术(杜邦RiboPrinter? System)的问世,大大简化了鉴定和分型流程,相比繁琐和对操作技术要求较高的PFGE技术具有显著的优势。
表四:Ribotyping技术用于肉毒梭菌及其他梭菌属菌株的分型能力分析

图二:肉毒梭菌EcoR I酶切的Ribotyping指纹图谱的聚类分析

图三:肉毒梭菌Hind III酶切的Ribotyping指纹图谱的聚类分析
自动化的Ribotyping技术已经在病原微生物的风险分析和关键点控制(HACCP)体系、GMP体系、食品和药品安全质控和质保、常规检测、环境监测、法规制定和基础研究等领域引起了广泛的关注并得到了长足的应用。
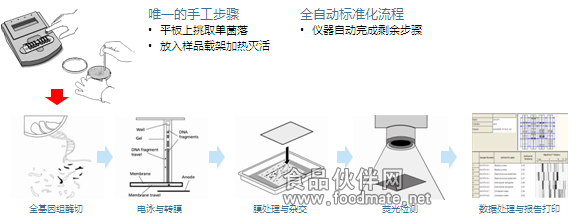
随着美国杜邦公司的RiboPrinter®系统的问世,Ribotyping技术实现了全自动化和标准化(见图四)。因其出众的性能、简单的操作,RiboPrinter®系统已经成为世界众多微生物学、分子生物学实验室开展微生物鉴定和分子分型的首选设备,具备如下特点:
1.高通量,检测速度快,鉴定加分子分型8小时内同步完成
2.结果准确,重复性和重现性好
3.广谱的鉴定和分型能力,未知菌株可直接上机,无需预检
4.通用试剂盒适用已知和未知细菌的鉴定和分型,及大规模筛查和应急检测需求
5.开放平台可满足更高特异性检测和个性化研究需求
6.支持种属特异试剂盒和多酶切方案满足更高分型力需求或建立菌株数据库
7.操作简单,自动化程度高,标准化流程内设,结果自动判读,非经验丰富人员也只需简单培训即可使用,节省不必要的用地和设备,人员8.培训更简单,成本更低
9.标准数据库容量为8528个基因模式,包括1741个种和297个属,标准菌株均来源ATCC(美国模式培养物研究所)、JCM(日本微生物菌种保藏中心)和DSMZ(德国微生物菌种保藏中心)。涵盖环境菌(如葡萄球菌、微球菌、芽孢杆菌、梭菌属等);致腐菌(芽孢杆菌、假单胞菌、明串珠菌、梭菌属等);致病菌(肠出血性大肠杆菌、沙门氏菌、金黄色葡萄球菌、志贺氏菌、单增李斯特菌、弧菌、弯曲杆菌、阪崎肠杆菌等);益生菌(乳酸杆菌、双歧杆菌、乳球菌、嗜热链球菌等)等多种类型
图四:杜邦RiboPrinter® System全自动微生物基因指纹鉴定系统及流程

Bruce等(1995 & 1996)和Webster等(1994)曾证明在沙门氏菌和单增李斯特氏菌菌株分型技术方面,RiboPrinter?系统比传统技术更为有效。
Wiedmann等(1996)曾成功用RiboPrinter®系统追溯到产品和环境中单增李斯特氏菌的污染,在动物源性病原菌疾病暴发的控制方面起到了积极作用。无独有偶,Bruce等(1996)也成功用RiboPrinter®系统追溯到了食品加工厂环境中的金黄色葡萄球菌污染。
在Hielm等人的研究基础上,Skinner等(2000)采用RiboPrinter®系统对30株肉毒梭菌和1株生孢梭菌的鉴定和分型能力进行了进一步的研究,获得了成功。
他们开展该工作的原因之一,是因为Hielm等(1998)曾报道,采用PFGE对非蛋白分解型肉毒梭菌进行分型比较困难,很难获得高质量的指纹图谱。而且在分析B、E、F三型非蛋白分解型肉毒梭菌时,菌株DNA的降解成了一个非常困扰的问题。而此前,Samore等(1996)已经报道了,在33株难辨梭菌(Clostridium difficile)的分型工作中,PFGE对其中的23株都没有成功分型,推测的原因也是菌株DNA的意外降解。
这31株各型的肉毒梭菌(见表五)均来自于美国食品药品监督管理局和罐头食品企业,被接种于改良的厌氧卵黄琼脂培养基上,在35℃下培养24h。用3mL缓冲液(2 mM Tris + 20 mM EDTA)淋洗平板及用灭菌接种针刮擦平板表面以获得营养细胞,制成30µL每管的细胞混悬液,直接用于RiboPrinter®系统分析,经系统自动的加热灭活、裂解、标准EcoR I酶切及后续分析等流程,获得了较为理想的结果。
表五:Skinner等用于分析的肉毒梭菌菌株表


31株菌的分型结果如表六和图五所示。Skinner等定义菌株相似度>93%的图谱被归为一个Ribogroup,故31株菌可被分成15个Ribogroup,并显示了较高的重现性。其中23株蛋白分解型菌株(含A型和B型),5株非蛋白分解型菌株(B型)和2株E型菌株及1株生孢梭菌均得到了高分辨的分型。
表六:31株肉毒梭菌和产气荚膜梭菌菌株经EcoR I酶切的Ribogroups模式列表
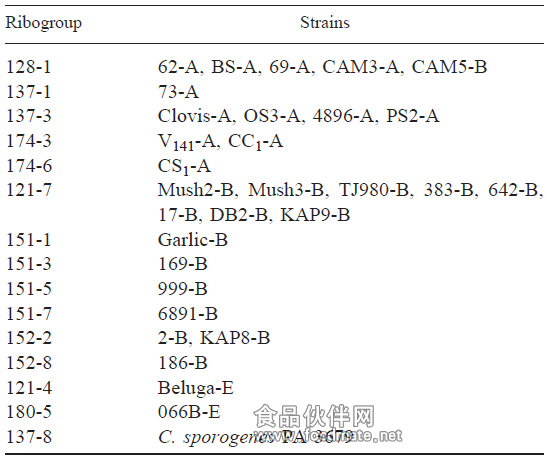
图五:30株肉毒梭菌经EcoR I酶切获得15组Ribogroup图谱

Skinner等随即对菌株的来源和菌株的分组进行了综合分析,证实了RiboPrinter®系统在菌株溯源方面的应用价值,见表七。
表七:部分肉毒梭菌菌株的追溯和图谱分析
Ribogroup | 菌株 | 来源与图谱分析 |
128-1 | CAM3-A, CAM5-B | 来源于同一个鸡肉蔬菜汤罐头加工厂,来自于同一个生产批次的两个罐头样本,分型结果在同一个Ribogroup |
137-3 | OS3-A, 4896-A | 均从1983年美国伊利诺伊州肉毒梭菌食物中毒事件中收集而来,其中4896-A来自病人的粪便,OS3-A来自于最终确认的原因食品——炒洋葱,这两株菌不仅被分在了一个Ribogroup中,且指纹图谱相似度高达99% |
121-7 | Mush2-B, Mush3-B | 均来自于1975年同一批次生产的鲜蘑菇,分型结果在同一个Ribogroup |
121-7 | 383-B, 642-B | 来源于1972-1973年生产的两个批次鲜蘑菇样本,分型结果在同一个Ribogroup |
137-8 | C. sporogenes PA 3679 | 该生孢梭菌的图谱与A型肉毒梭菌的相似,从细菌分类学上很容易得到解释,因为其与组I肉毒梭菌具有相似的培养和生化特征,唯一的区别是不会产BoNTs |
从表七中可以看出,来源相近的肉毒梭菌菌株最终被追溯到同一个Ribogroup组别(如128-1, 137-3, 121-7,),而生化性质与肉毒梭菌接近的生孢梭菌则被分为了一个新的Ribogroup组别(如137-8),体现了RiboPrinter®系统较好的菌株追溯能力,及干扰菌株的排他能力,这在食物中毒病原体追溯、原因食品追溯、流行病学调查、院内感染、生产环境监测和污染源追溯等领域有十分重要的应用价值。
图六:29株肉毒梭菌经EcoR I酶切获得的9组Ribogroup图谱

Kennett等(2006)也采用RiboPrinter®系统及EcoR I酶切方案,对梭菌属的7种,共计49株可引发食源性疾病的梭菌菌株进行了鉴定和分型,包括:耐氧梭菌(Clostridium aerotolerans),拜氏梭菌(Clostridium beirjerinckii),腐败梭菌(Clostridium putrificum),肉毒梭菌,酪酸梭菌,产气荚膜梭菌和生孢梭菌。Kennett等同时设计了EcoR V酶切方案消化17株肉毒梭菌,以考察不同酶切方案对鉴定和分型效果的改善作用。结果证实RiboPrinter®系统可以较好地鉴定和分型这些梭菌属的菌株,还可通过调整限制性酶切方案来分型不同表型和产毒型的菌株,可选的限制性内切酶除了EcoR I和EcoR V之外,还可选Cla I,Hind III,Spe I,Rsa I等,部分图谱可见图六。
综上所述,杜邦RiboPrinter® System在肉毒梭菌及其他梭菌属细菌的鉴定和分子分型领域有着广阔的应用前景和突出的使用价值。依托杜邦的行业领先技术,该鉴定分型产品的技术路线可有效地保障政府检测机构、第三方检测实验室、研究院所和食品、药品企业快速、准确、标准化地鉴定和分型肉毒梭菌,建立菌株信息库,有效追溯污染菌株,从而预警和控制潜在的肉毒梭菌暴发和致命食源性疾病发生的风险。














